Las comunidades microbianas de la viña (suelo y vid) juegan un papel importante en la productividad y resistencia a enfermedades de la vid. Además, estos microorganismos nativos están también presentes en los tanques de fermentación, formando parte del proceso de la transformación del mosto en vino y de sus características organolépticas.
Es de especial interés por tanto el conocer, conservar y promover la riqueza microbiana asociada al cultivo del Txakoli, actualmente poco estudiada. Un mayor conocimiento de la estructura y composición microbiana del viñedo, así como los factores bióticos y abióticos que lo modulan, permitirán al viticultor actuar para mejorar el estado sanitario de su viña, y ayudará a poseer un mayor control del proceso de vinificación, asegurando en definitiva un vino de mayor calidad.
Objetivos
La llamada metagenómica –el estudio de la microbiota de muestras ambientales (suelo, uva, etc) sin cultivarlos ni aislarlos– ha comenzado recientemente a utilizarse en los sistemas agrícolas y está demostrando ser una aproximación metodológica idónea para lograr una mejor comprensión de la complejidad de las interacciones entre microorganismos–planta.
Siendo la comunidad microbiana básica para la mejora de la producción y la salud del viñedo, la finalidad de esta línea de investigación es utilizar aproximaciones metagenómicas para establecer un marco de referencia para la conservación y promoción de la diversidad microbiana asociada al Txakoli.
Trabajamos en dos líneas de actuación:
- Estudio 1: La caracterización de la trayectoria de los microorganismos nativos de la vid desde la cosecha a la bodega: monitorización de la fermentación.
- Estudio 2: La caracterización del perfil taxonómico y metabólico de la variedad de uva Hondarrabi zuri en las tres regiones productoras de Txakoli.
Investigación
Estudio 1: Caracterización de la trayectoria de los microorganismos nativos de la vid desde la cosecha a la bodega: monitorización de la fermentación
En este trabajo se empleó la secuenciación masiva para contribuir al conocimiento sobre la diversidad y composición bacteriana asociada a la uva y fermentos de Txakoli, así como para determinar la influencia de la bodega, la variedad de uva, la etapa de fermentación y la levadura inoculada en su riqueza y abundancias/distribución.
Muestras
En la añada de 2015, se recogieron 123 muestras de 8 bodegas situadas en varias zonas de la D.O. Bizkaiko Txakolina.
Factores estudiados:
- 4 variedades de uva: Hondarrabi zuri, Hondarrabi beltza, Petit Courbu, Pinot noir.
- Distintos Estadios: Mosto tras prensado, desfangado, fermentación alcohólica (inicio/mitad/final).
- Elaboraciones con distintas levaduras industriales: ICV okay, Ysseo cross evolution, Biodiva cross, Exot, Ecológica neutra, fermentación espontánea.
- Experimento en viña con adición o no de precursores aromáticos
Resultados más relevantes: (Trabajo Fin de Máster)
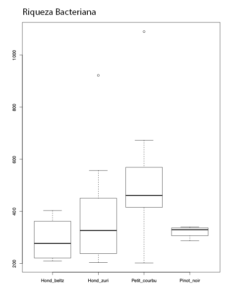
Riqueza= Diversidad=Número de especies distintas
- Se ha detectado una media de 300 bacterias distintas en las muestras estudiadas.
- La riqueza bacteriana es mayor en el mosto y disminuye durante la fermentación, ya que el grado de alcohol y la cada vez mayor presencia de levaduras impiden su desarrollo, volviendo a aumentar al final de la fermentación alcohólica, cuando la competencia es menor.
- Petit courbu es la variedad con mayor diversidad bacteriana (con una media de riqueza cercana a 425 bacterias distintas en mosto).
Composición=distribución de las abundancias
- La asignación taxonómica de las secuencias mostró que el Orden más abundante es el de Sphingomonadales, seguido de Rhizobiales (ambos de la Clase Alphaproteobacteria y del Phylum Proteobacteria). Entre estos dos órdenes suman el 85,3% del total de los microorganismos.
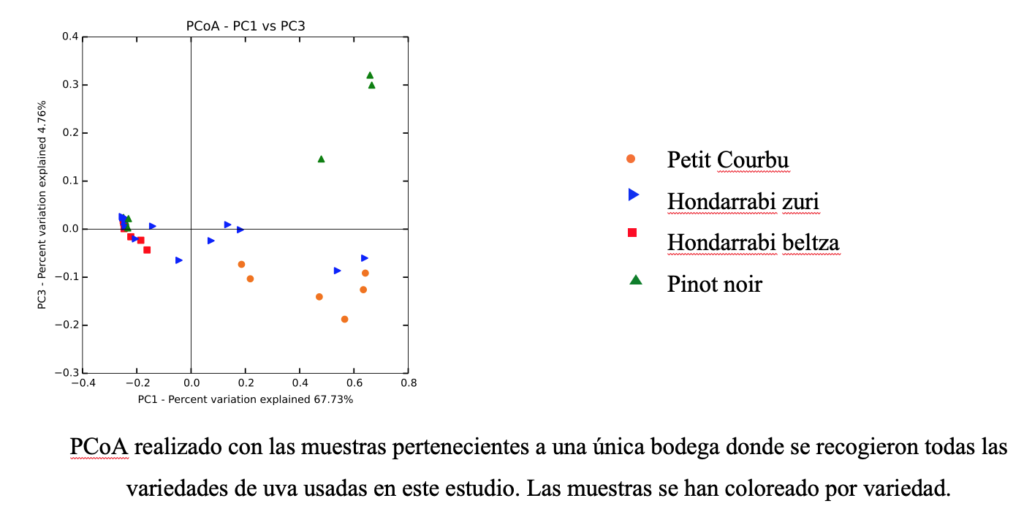
- La Pinot noir es la variedad con composición bacteriana más diferenciada: las abundancias de las bacterias presentes en esta variedad son distintas a las halladas en H. zuri, Petit courbu y H. beltza. Esto puede deberse en este caso al menor parentesco genético de Pinot noir al resto de las variedades estudiadas. La Petit courbu muestra también un perfil bacteriano distinto.
- Se encuentran diferencias en la composición bacteriana entre bodegas, lo que es debido en parte, a los distintos protocolos de cada una (distinto momento de adición del SO2, la adición o no de nutrientes…).
- La fermentación espontánea permite la proliferación de una mayor diversidad de bacterias, y entre otras, la sobre-representación del género Gluconobacter, tradicionalmente asociado al deterioro del vino por producir picado ácido, y por tanto es un grupo especialmente importante a monitorizar si se realiza este tipo de fermentaciones.
- Las elaboraciones inoculadas con una levadura con fenotipo killer neutra (como la eco neutra en este estudio) permiten la proliferación de mayores abundancias de Weissella (bacteria láctica heterofermentativa) y Geobacillus.
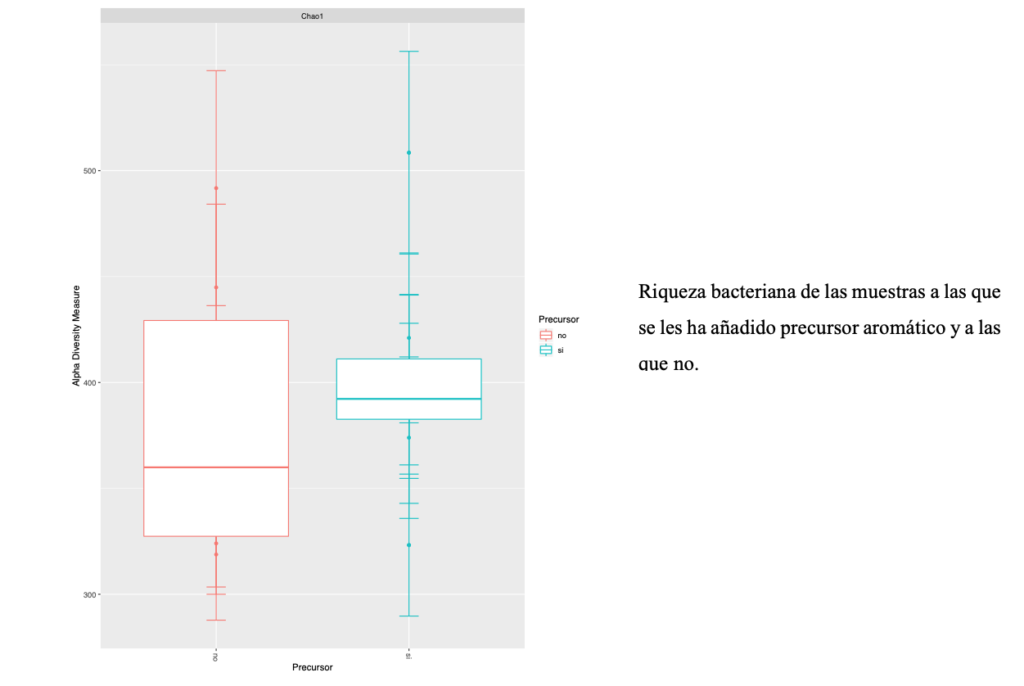
- El aporte de precursores aromáticos como LalVigneä Aroma en viña aumenta la riqueza bacteriana. El análisis metabólico sugiere ligeras diferencias en la composición química entre las muestras a las que se les añadió o no el precursor en viña, únicamente apreciable en las etapas de mitad y final de la fermentación alcohólica.
Estudio 2: Caracterización del perfil taxonómico y metabólico de la variedad de uva Hondarrabi zuri en las tres regiones productoras de Txakoli
En este trabajo se ha empleado la secuenciación masiva para contribuir al conocimiento sobre la diversidad y composición bacteriana asociada a viñedos de H. zuri y pretende:
- Determinar el grado de distinción de las parcelas/bodegas/regiones/DOs para definir su terroir microbiano mediante el estudio comparativo de sus perfiles de bacterias y hongos/levaduras (Metagenómica) y metabólitos (H-RMN).
- Crear un inventario de organismos beneficiosos y perjudiciales, para determinar el estado de salud de la viña mediante el estudio de la presencia/abundancia de microorganismos patógenos, antagonistas y promotores de crecimiento.
Muestras:
Se recogieron 82 muestras de suelo y 210 muestras de uva (Hondarrabi zuri) anuales, recopiladas en 23 bodegas distribuidas en las 3 Denominaciones de Origen del País Vasco: 15 bodegas en Bizkaia (27 parcelas), 5 bodegas en Gipuzkoa (7 parcelas) y 3 bodegas en Araba (8 parcelas).
Factores estudiados:
- Experimentales: DO, bodega, viñedo y año.
- Asociados al viñedo: orientación, pendiente, altitud, coordenadas geográficas, sistema de conducción (parral/espaldera), portainjerto (41B, 3309, SO4, Gravessac, Pie franco).
- Edáficos del suelo: pH, conductividad, humedad, Carbono total y Nitrógeno.
- Química del mosto: pH, acidez total, alcohol probable, Nitrógeno asimilable.
- Climáticos: temperatura total, precipitación total.
Para ver una explicación general del trabajo e interpretar los gráficos de resultados ver video explicativo.
Resultados más relevantes:
COMUNIDAD BACTERIANA EN SUELO:
Riqueza:
- Los suelos estudiados presentan un amplio rango de pH (4.21 – 8.34).
- Albergan entre 375 y 558 bacterias distintas, y esta riqueza varía entre las parcelas estudiadas.
- Los suelos más pobres en bacterias son, en general, aquellos con pH bajos (pH 4-5), mientras que las parcelas de mayor riqueza se corresponden con suelos con pH de alrededor de 6.6-7, disminuyendo levemente después. Estos resultados son consistentes con el rango de pH óptimo del suelo estimado para H. zuri (pH 6.5 – 7.0).

Nomenclatura de las parcelas: Unestablished= parcela de viñedo recien plantado; Las parcelas que comienzan por B pertenecen a la DO de Bizkaia, con G a la DO de Gipuzkoa y con A a la DO de Araba.
Composición:
- Proteobacteria es el filo dominante en los suelos de Txakoli (30,27% de abundancia relativa), seguido por Acidobacteria (16,8%), Verrucomicrobia (16,5%) y Planctomycetes (9,2%). Composición microbiana por viñedos.
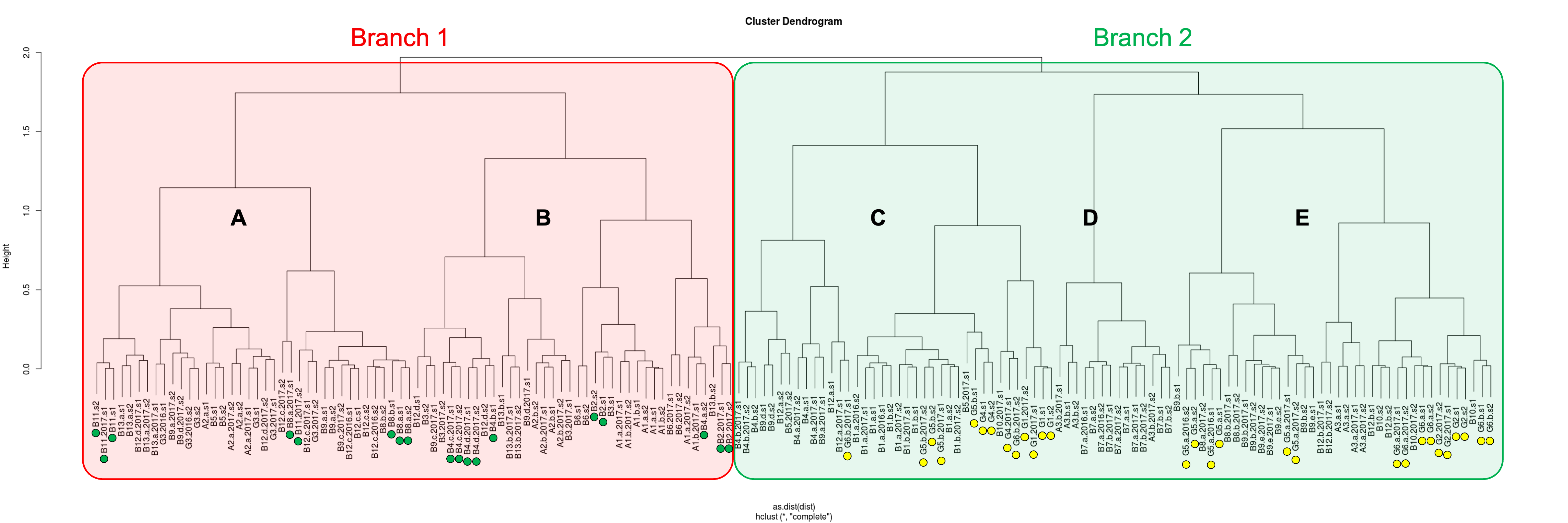
- El pH del suelo determina, en parte, la composición bacteriana hallada. Las 82 parcelas estudiadas se agrupan en dos grandes grupos que difieren en la abundancia de bacterias particulares relacionadas con el ciclo del nitrógeno y del carbono.
- No se encuentra un perfil bacteriano específico de DO.
Si bien todas las parcelas de Bizkaia situadas en la costa (puntos verdes) se agrupan en el branch 1, las de interior se distribuyen en ambos grupos, al igual que las parcelas de Araba. En cuanto a las de Gipuzkoa, todas aquellas costeras (con punto amarillo) se sitúan en el branch 2, mientras que la única parcela estudiada en el interior se agrupa en el branch 1.
COMUNIDAD BACTERIANA EN UVA:
Riqueza:
- Las uvas H. zuri albergan de 100 a 450 bacterias distintas (aprox. 180 de media) y hay diferencias entre parcelas.
- El portainjerto es el factor que mayormente determina las diferencias en riqueza bacteriana encontradas entre parcelas. Las vides en patrón 41B y Gravessac muestran una mayor riqueza, mientras aquellas en SO4 muestran valores de diversidad algo menores.

Composición:
- Sphingomonas (44%) y Methylobacterium (18%) son los géneros con mayor abundancia relativa en uva, seguidos por Flavobacteriales (1.7%), Lactobacillales (1.5%), Actinomycetales (1.6%) y Neisseriales (1.2%). Composición microbiana por viñedos.
- La presencia/ausencia y abundancias de bacterias varía entre añadas (debido a diferencias climáticas), pero las similitudes/diferencias entre parcelas se mantienen
- No se encuentra un perfil bacteriano específico de DO.
- La composición bacteriana de la uva está determinada por el portainjerto, respaldando la idea de que el genotipo de las raíces es clave para el reclutamiento selectivo de microorganismos que luego colonizan los compartimentos aéreos (hoja, fruto, uva) en planta.
Microorganismos cuya abundancia se ve aumentada por portainjerto:
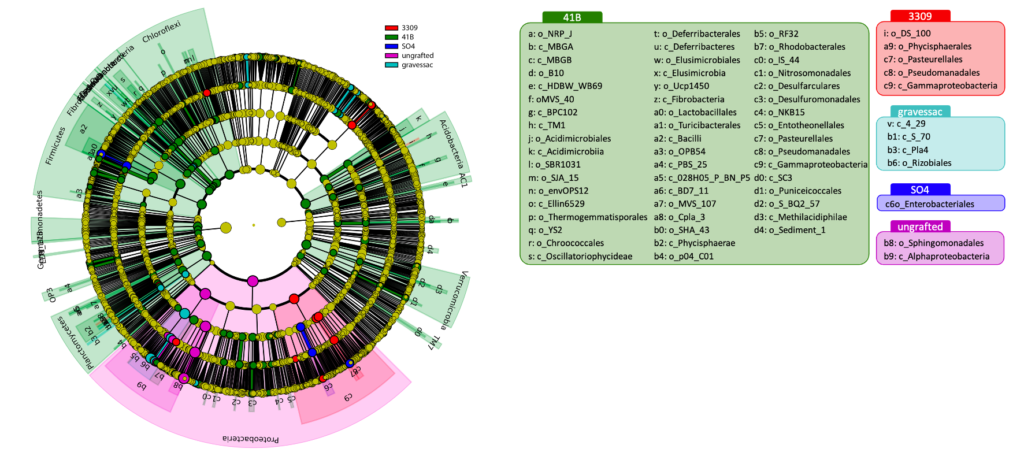
41B
Muestran un aumento de Plactomycetes, Nitrospira y Deltraproteobacteria, involucradas en procesos de nitrificación y fijación de nitrógeno. Gluconobacter, miembro clave del mosto y el vino, también se ve enriquecido en uvas con este portainjerto.
3309
Muestran mayores abundancias de Curtobacterium y Pseudomonadales, antagonistas frente a enfermedades fúngicas, y por tanto sugieriendo que la H. zuri injertada en este patrón podría mostrar mayor resitencia a dichos patógenos.
Gravessac
Muestran mayores valores de Methylobacterium y Phylobacterium, bacterias generalmente encontradas en la superficie de la uva, pero que son capaces de sobrevivir durante la fermentación contribuyendo a las características finales del vino.
SO4
Muestran un enriquecimiento de Acinetobacter rhizosphaerae (Pseudomonadales), conocidas rizobacterias promotoras del crecimiento. Además esta uva contiene mayores abundancias de Lactobacillus, principales agentes de la fermentación maloláctica, pero que en ocasiones pueden tener consecuencias negativas para la calidad de los vinos, dando lugar a picado láctico.
Pie Franco
La uva sin portainjerto muestra enriquecimiento de Agrobacterum vitis y A. tumefaciens (Alphaproteobacterias), causante de la enfermedad “agalla de corona”, lo que sugiere que estas vides están menos protegidas frente a esta enfermedad.
Difusión
- 2017: Entrevista de Iratxe Zarraonaindia y Elena Garcia en el programa de TV TEKNOPOLIS: “Genética avanzada para mejorar el txakoli” (Fundación Elhuyar)
- 2017: Entrevista de Iratxe Zarraonaindia el SER Euskadi, programa “I al Cuadrado” Controlar ordenadores con la mirada y microorganismos invisibles”
- 2018: Presentación Oral de Igor Baroja en el congreso : VIII Congreso Ibérico de las Ciencias del Suelo, Donostia San-Sebastian, 2018. Presentación oral
- 2019: Charla Divulgativa en el Pint of Science Festival de Elena Garcia e Iratxe Zarraonaindia titulada “El dinamismo del txakoli”
- 2021: Presentación de Poster de Igor Baroja en Macrowine 2021
Financiación
- Grupos consolidados IT558 -10. Entidad Financiadora: Consejería de Educación, Universidades e Investigación, Gobierno Vasco.
- Ayudas I+D+I de los sectores agrario, alimentario y pesquero de la CAPV. Título del Proyecto: Metagenómica del Txakoli: hacia una vitivinicultura personalizada. 10/2016 – 31/12/2018. Entidad Financiadora: Departamento de Desarrollo Económico y Competitividad del Gobierno Vasco.
- Beca predoctoral concedida a Igor Baroja en el Programa Predoctoral de Formación de Personal Investigador No Doctor del Departamento de Educación del Gobierno Vasco. (2016- 2020).
Participantes
- D.O. Bizkaia
- D.O. Getaria
- D.O. Araba
- Ardoatek
- Asociación de txakolineros de Bizkaia – BIALTXA